Инструкция Swiss PDB Viewer презентация
Содержание
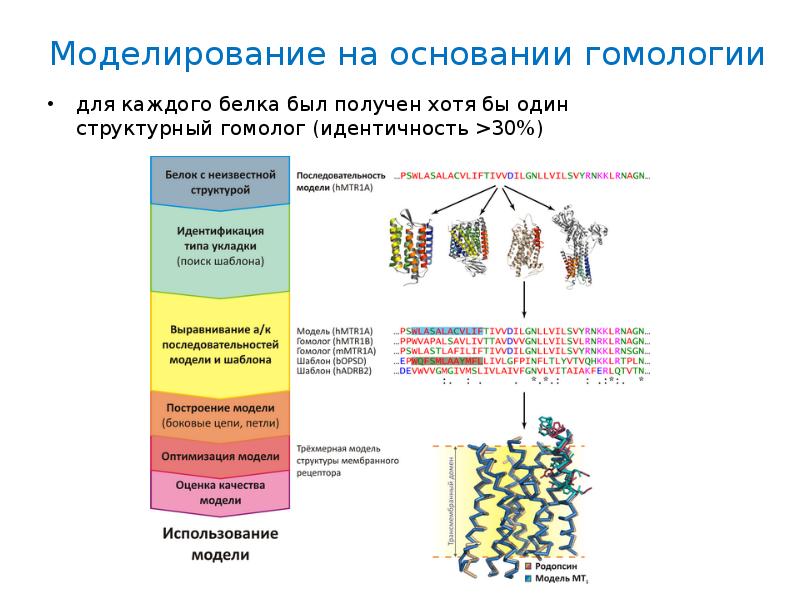
- 2. Моделирование на основании гомологии для каждого белка был получен хотя бы
- 3. Homology modeling Моделирование неизвестной структуры одного белка, на основании его гомологичности
- 4. ExPASY (Expert Protein Analysis System) Экспертная система анализа белков – протеомный сервер Института
- 5. Подходы: Полностью автоматическое выравние последовательности шаблона и модели. Первичная последовательность белка
- 6. http://www.expasy.org/swissmod
- 7. Optimise (Project) mode 1) Загрузка файла моделируемого белка: File>Import>Grab from server
- 8. Создание проекта моделирования Если загружается несколько образцов: Fit>Iterative Magic Fit Fit>Fit
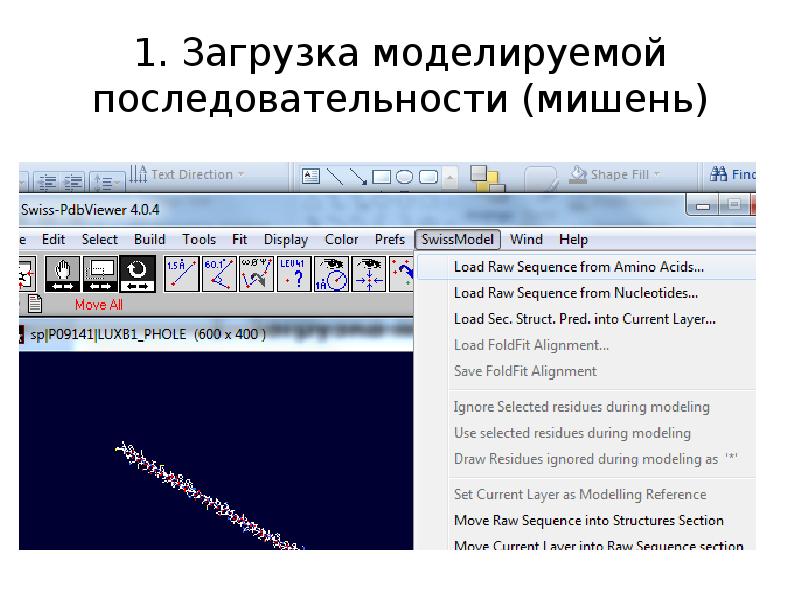
- 9. Loading a target sequence Загрузка последовательности моделируемого белка FastA: SwissModel>Load Raw
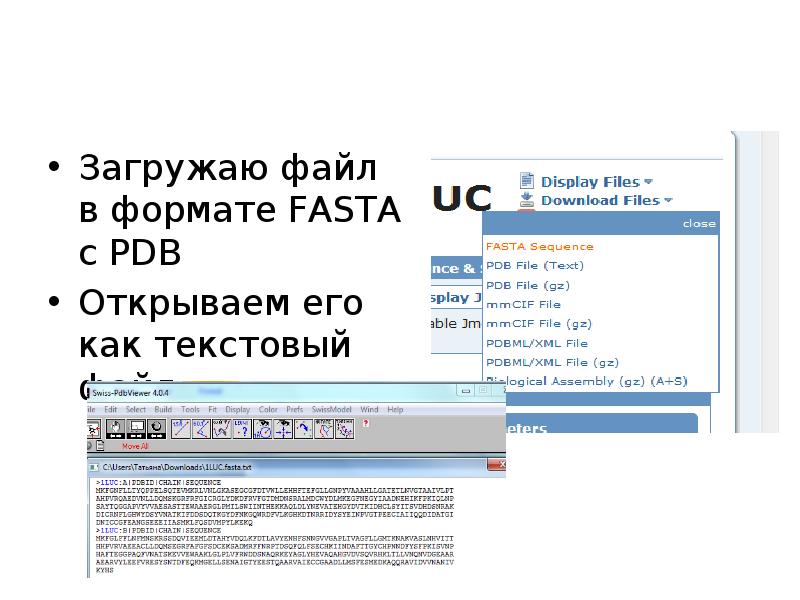
- 10. Загружаю файл в формате FASTA с PDB Открываем его как
- 11. 1. Загрузка моделируемой последовательности (мишень)
- 12. 2.Поиск гомологичной последовательности (шаблона) Выделяем все молекулы мишени Edit>BLAST Selection vs.
- 13. 3.Создание проекта модели Управление выравниванием между последовательностями модели и шаблона Будет
- 14. 4.Выравнивание последовательности мишени по шаблонам Fit>Fit Raw Sequence Если загружено более
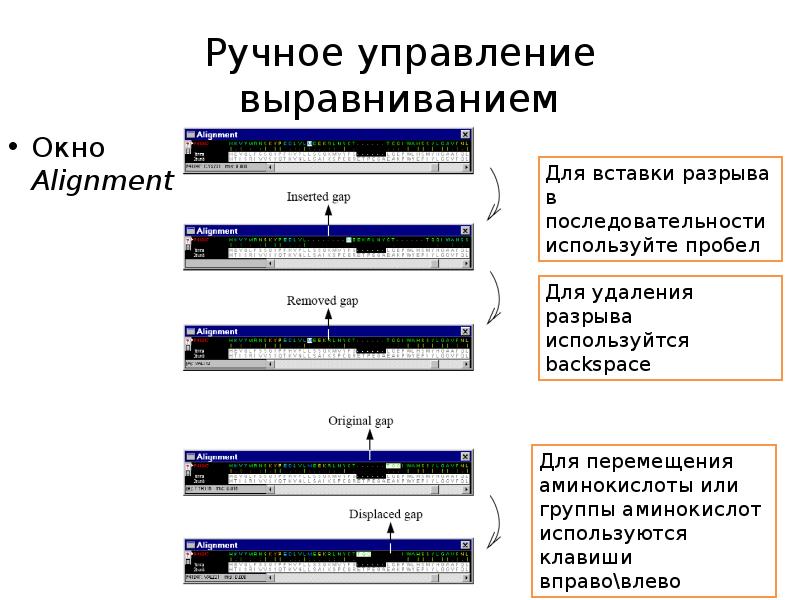
- 15. Ручное управление выравниванием Окно Alignment
- 16. Для наиболее удобного управления используйте окно Graphic – разрыв в последовательности
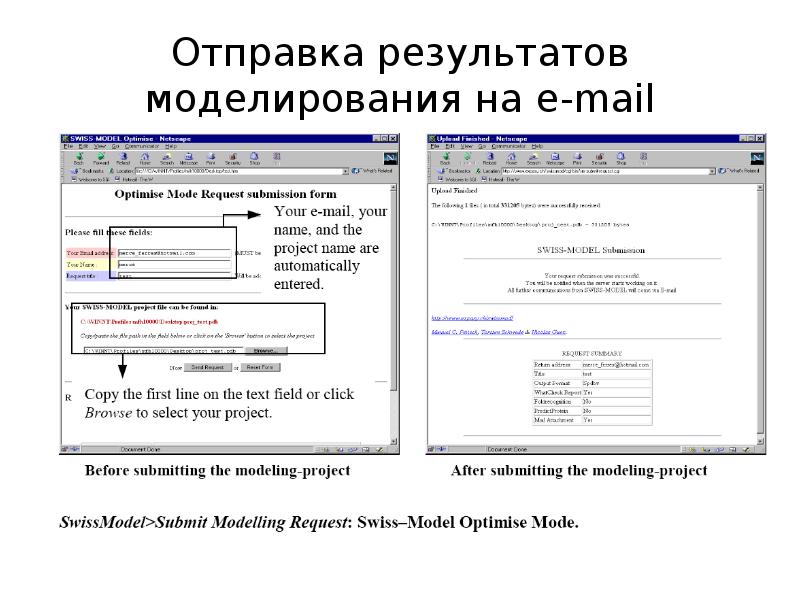
- 17. Отправка результатов моделирования на e-mail
- 18. Cовершентствование модели Построенная 3D модель будет отправлена на почту в виде
- 19. В зависимости от качества модели: Незначительная корректировка боковых цепей:
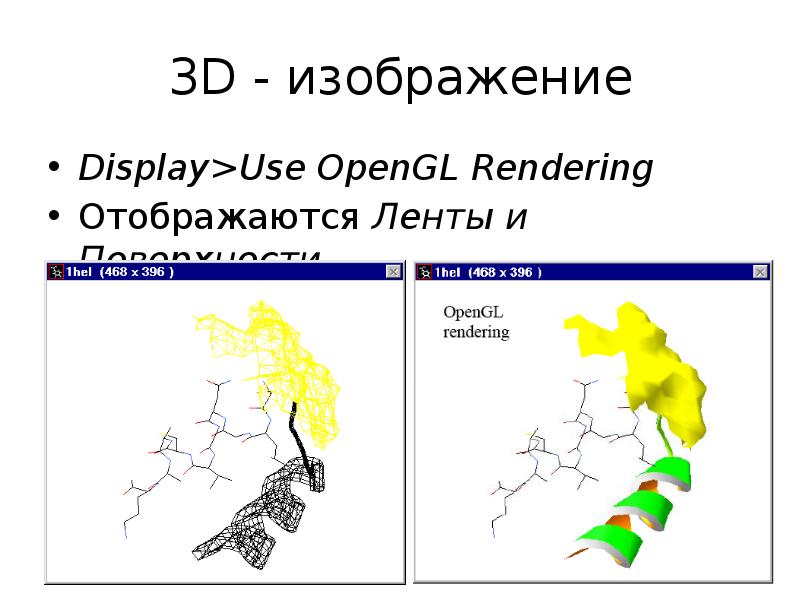
- 20. 3 типа изображения
- 21. 3D - изображение Display>Use OpenGL Rendering Отображаются Ленты и Поверхности
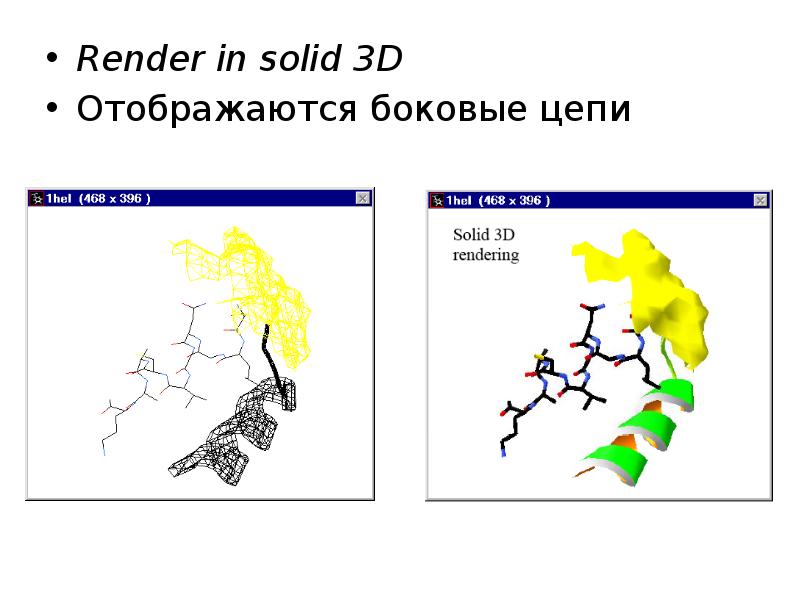
- 22. Render in solid 3D Render in solid 3D Отображаются боковые цепи
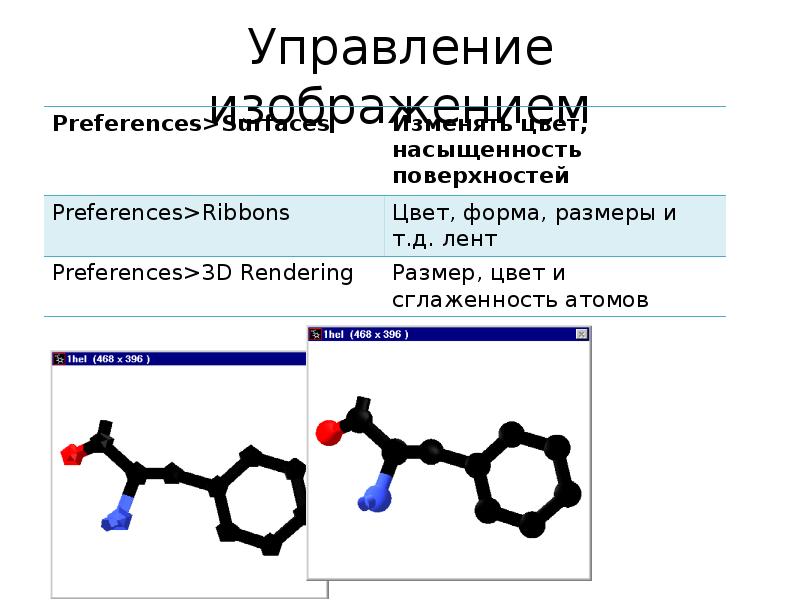
- 23. Управление изображением
- 24. Стерео изображение Display>Stereo View red and blue
- 25. Скачать презентацию















Слайды и текст этой презентации
Похожие презентации





























