Молекулярные базы данных. Принцип действия и характеристики основных компьютерных программ презентация
Содержание
- 2. Новейшим методом изучения природных молекул является применение информационных систем. Новейшим
- 3. Компьютерным моделированием молекулярно-генетических и смежных процессов занимаются такие науки как биоинформатика,
- 4. Биоинформатика Биоинформатика — это область науки, разрабатывающая и применяющая вычислительные алгоритмы
- 5. Биоинформатика включает в себя: Биоинформатика включает в себя: базы данных, в
- 6. Задачи, решаемые биоинформатикой
- 7. Биолог в биоинформатике обычно имеет дело с базами данных и инструментами
- 8. Второй тип – курируемые базы данных, за достоверность которых отвечает хозяин
- 9. Поддержание базы требует работы кураторов или аннотаторов. Тем не менее, даже в
- 10. Третий тип – производные базы данных. Такие базы получаются в результате
- 11. И интегрированные базы данных, в которых вся информация (курируемая, не курируемая)
- 12. Теперь перейдем к рассмотрению инструментов биоинформатики. Инструменты определяются задачами, которые мы
- 13. Как сравнивают последовательности? Запишем одну последовательность под другой: Как сравнивают последовательности?
- 14. Таким образом, первым делом после секвенирования последовательности ищут в базах данных
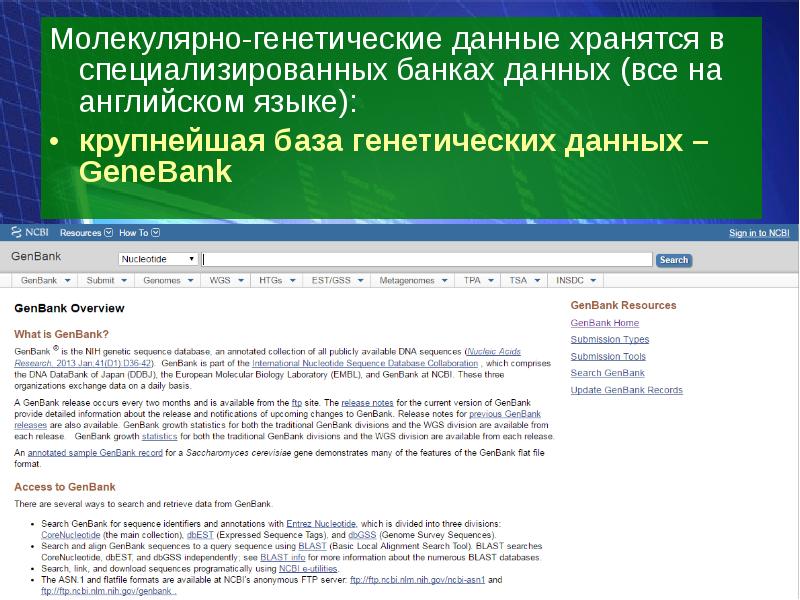
- 15. Молекулярно-генетические данные хранятся в специализированных банках данных (все на английском языке):
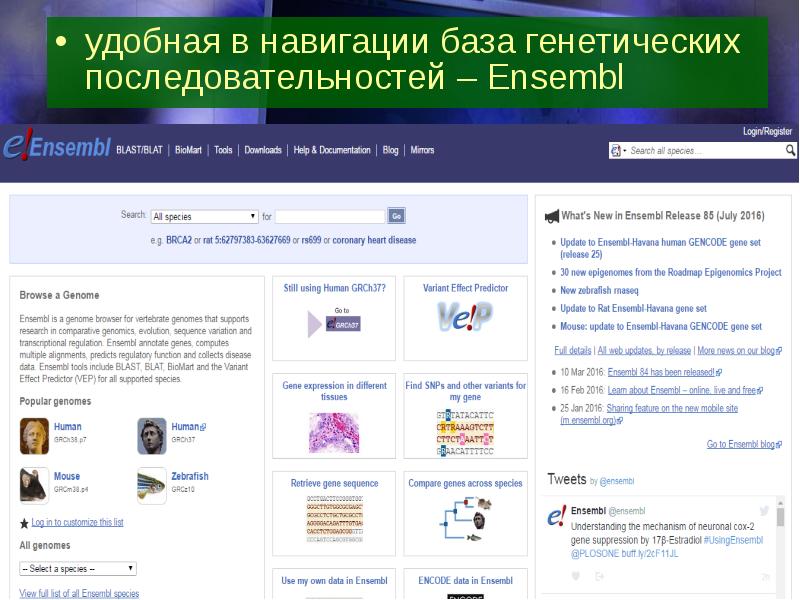
- 16. удобная в навигации база генетических последовательностей – Ensembl удобная в
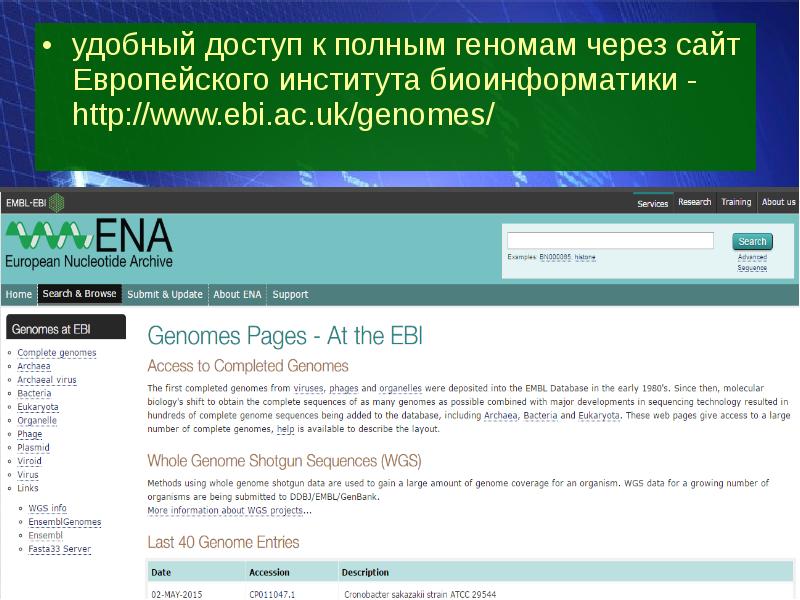
- 17. удобный доступ к полным геномам через сайт Европейского института биоинформатики -
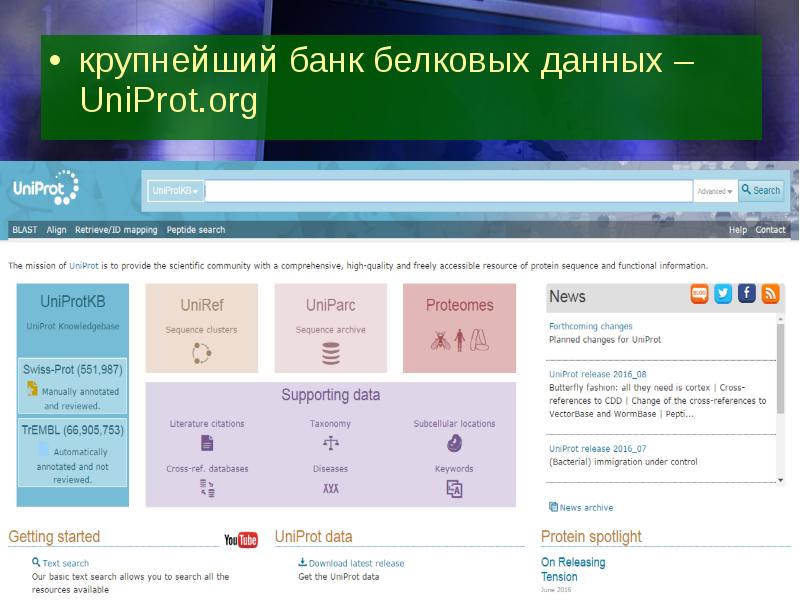
- 18. крупнейший банк белковых данных – UniProt.org крупнейший банк белковых данных
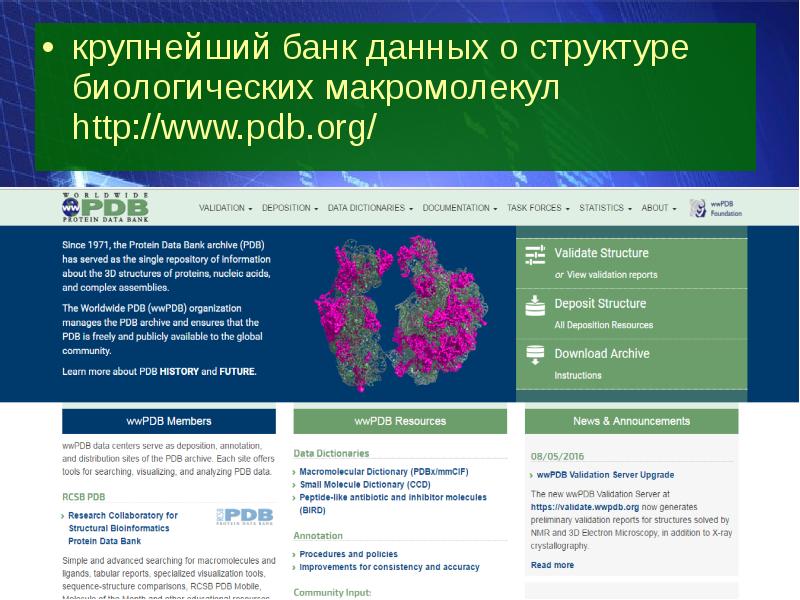
- 19. крупнейший банк данных о структуре биологических макромолекул http://www.pdb.org/ крупнейший банк
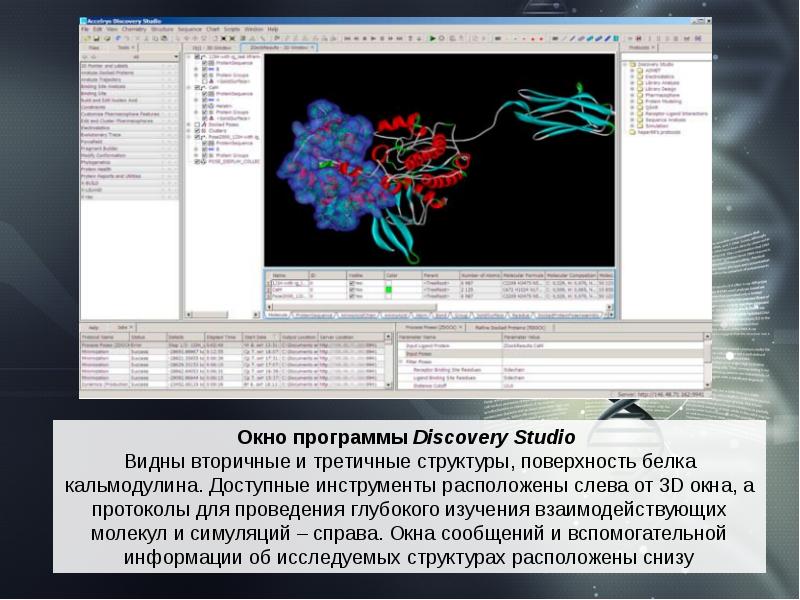
- 20. Информационные системы, касающиеся моделей макромолекул и надмолекулярных структур: GeneBank &
- 22. CAZy: Carbohydrate-Active Enzymes Database. На сайте представлена современная классификация ферментов синтеза
- 23. http://molbiol.edu.ru/review/01_01.html
- 29. Форматы файлов, используемых в биоинформатике FASTA >roa1_drome Rea guano receptor type
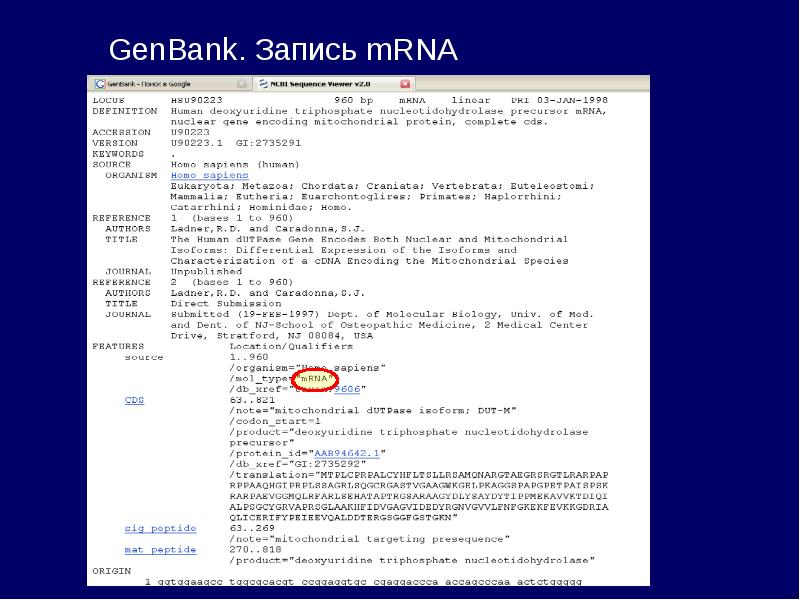
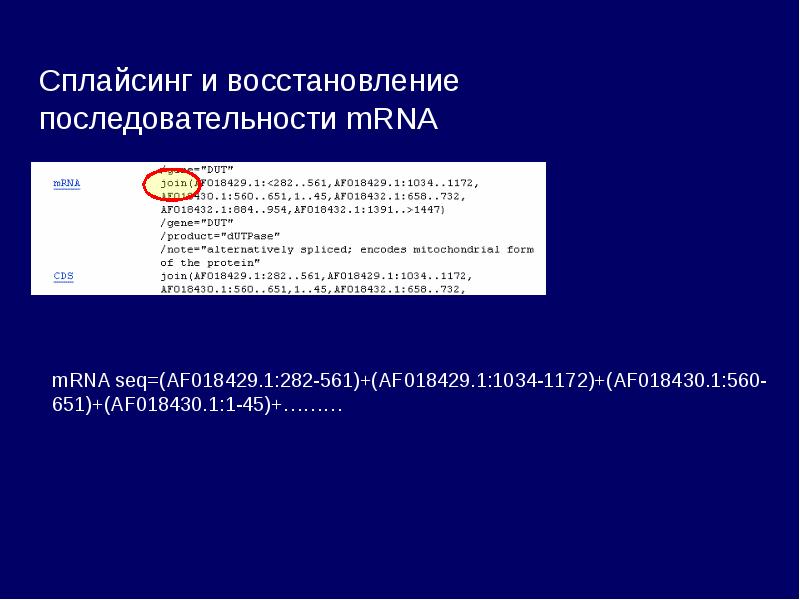
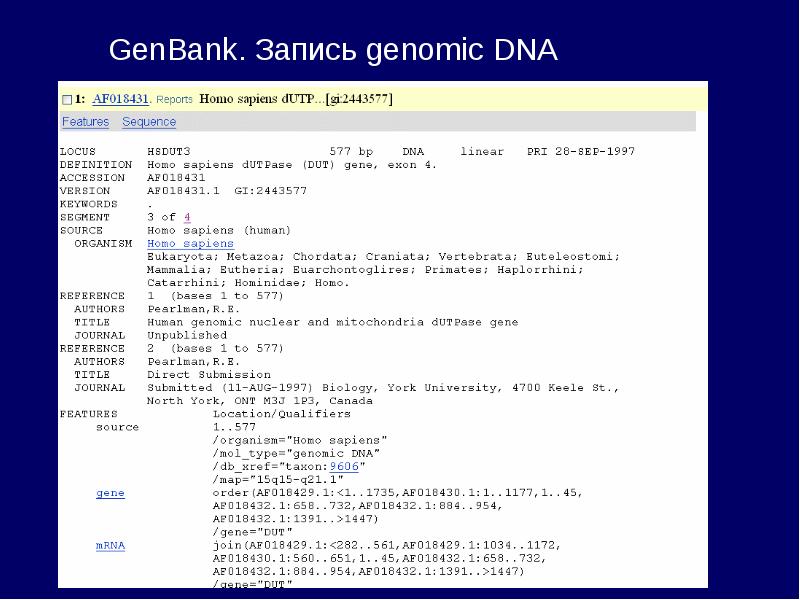
- 30. GenBank GenBank
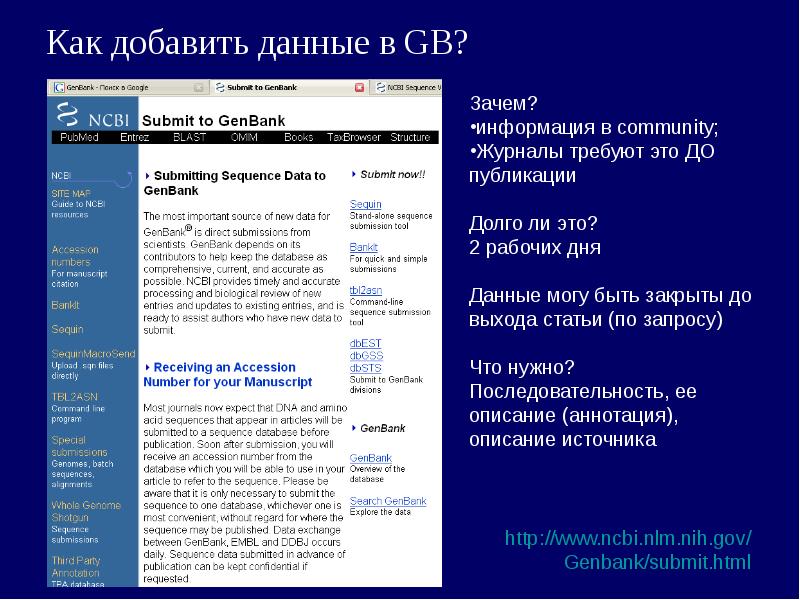
- 36. Как добавить данные в GB?
- 37. Форматы описания белков PDB PDB-XML MMDB-Cn3D
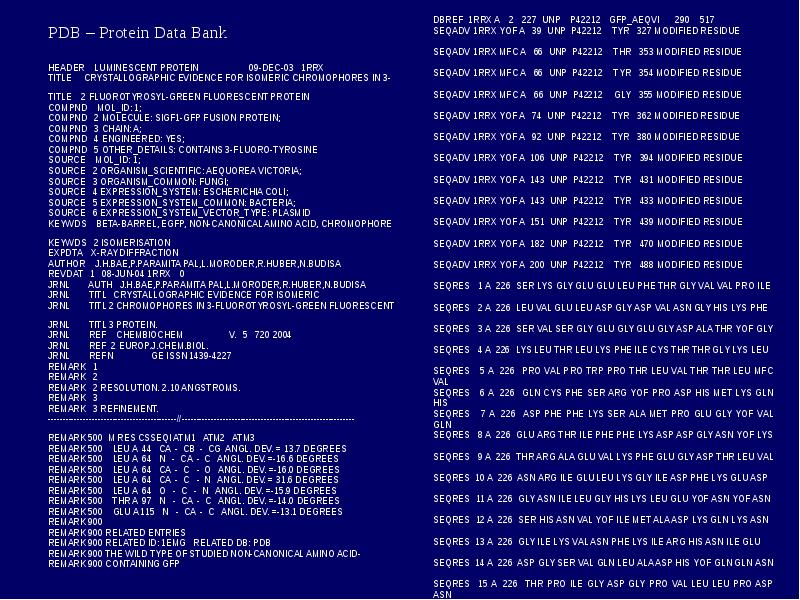
- 38. PDB – Protein Data Bank PDB – Protein Data Bank HEADER
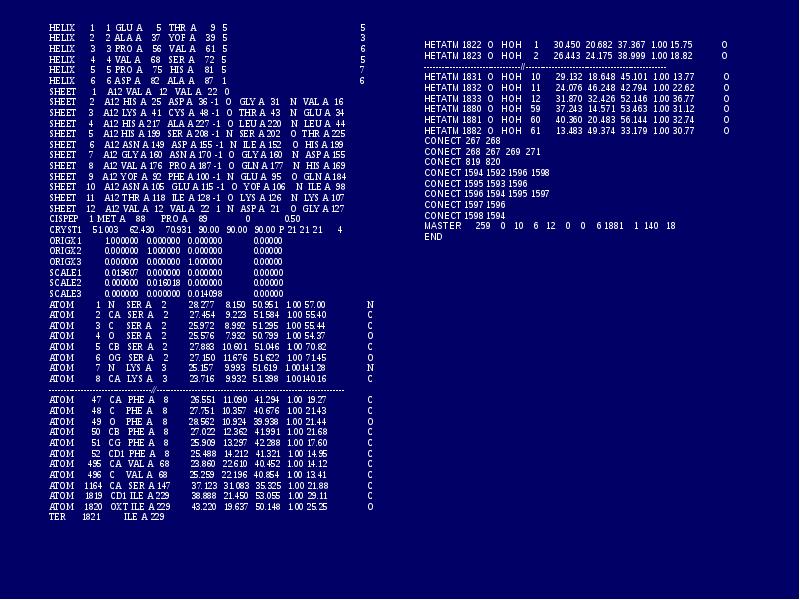
- 39. HELIX 1 1 GLU A 5 THR A 9 5
- 40. PDB-XML PDBML: the representation of archival macromolecular structure data in XML.
- 41. Спасибо за внимание! Спасибо за внимание!
- 42. Скачать презентацию




























Слайды и текст этой презентации
Скачать презентацию на тему Молекулярные базы данных. Принцип действия и характеристики основных компьютерных программ можно ниже:
Похожие презентации